Kit FastKing RT (con gDNasi)
Caratteristiche
■ Alta efficienza: l'enzima FastKing RT è modificato con un motivo idrofobo, con un'efficienza RT superiore al 95%.
■ Sensibile: i modelli a partire da 1 ng possono essere identificati con precisione.
■ Resistenza: Capace di trascrizione inversa di modelli complessi, con perfetta resistenza alle impurità.
■ Flessibile: la rimozione del DNA genomico e la trascrizione inversa sono state completate separatamente. I primer sono stati miscelati separatamente in un tubo, in modo flessibile per cambiare altri primer.
Specifiche
Tipo: trascrittasi inversa modificata dal gene, gDNasi
Procedure: due fasi (rimozione del DNA genomico e RT)
Efficienza RT: >95%
Modello: 1 ng- 2 μg
Tempo di funzionamento: ~21 min
Applicazioni: Il cDNA retrotrascritto può essere utilizzato nella PCR convenzionale, nella PCR in tempo reale, nella costruzione di librerie di cDNA.
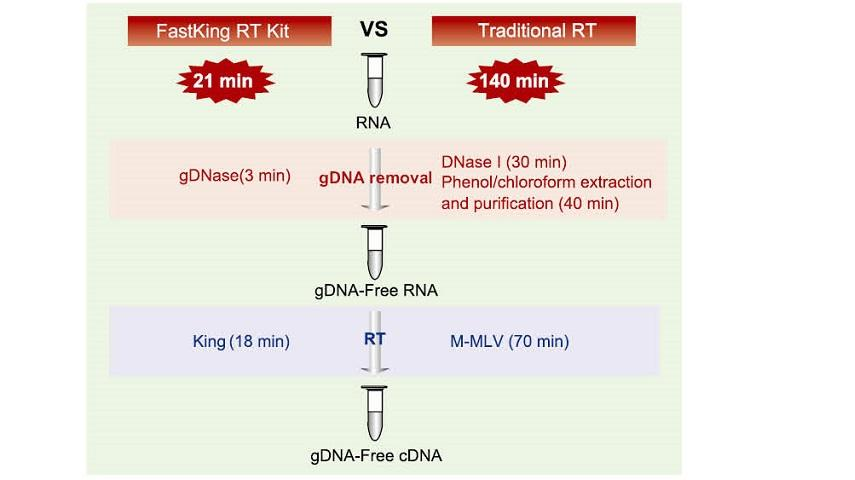
21 min di reazione in un tubo
Sono necessari solo 21 minuti per completare la rimozione del gDNA e l'efficiente processo di trascrizione inversa nello stesso tubo senza sostituire il tubo di reazione e il processo di trattamento indipendente della DNasi I. Rispetto al metodo tradizionale che richiede un'operazione in 12 fasi e una reazione di 140 minuti, semplifica notevolmente le fasi operative e consente di risparmiare molto tempo operativo.
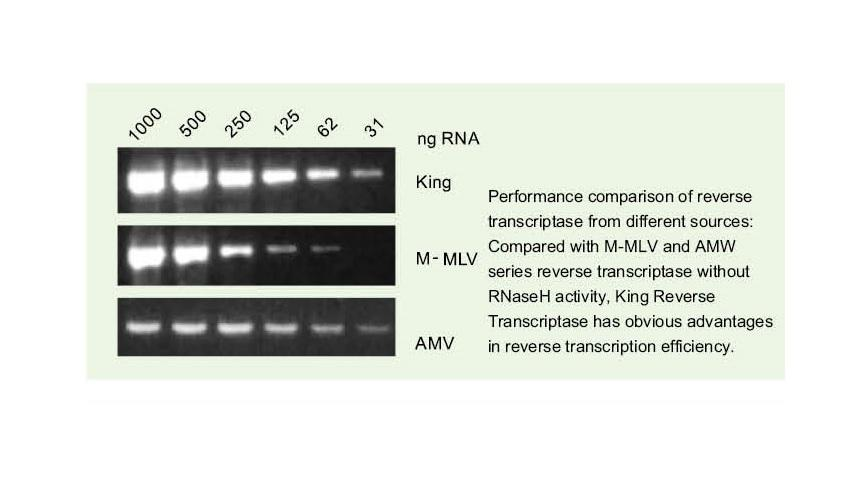
Eccezionale qualità di King RTase
——Efficienza di trascrizione inversa ultraelevata
——L'efficienza della trascrizione inversa è superiore al 95%
La trascrittasi inversa generale ha un'efficienza di trascrizione inversa del 40-60% e la resa di cDNA può essere aumentata di una maggiore quantità di carico di RNA. La trascrittasi inversa King può raggiungere un'efficienza di trascrizione inversa superiore al 95% grazie alla sua elevata affinità unica per i modelli di RNA. Pertanto, gli esperimenti successivi possono essere soddisfatti senza la necessità di una grande quantità di input di RNA, che consente di risparmiare RNA e consente un'elevata purezza e un'elevata resa di cDNA.
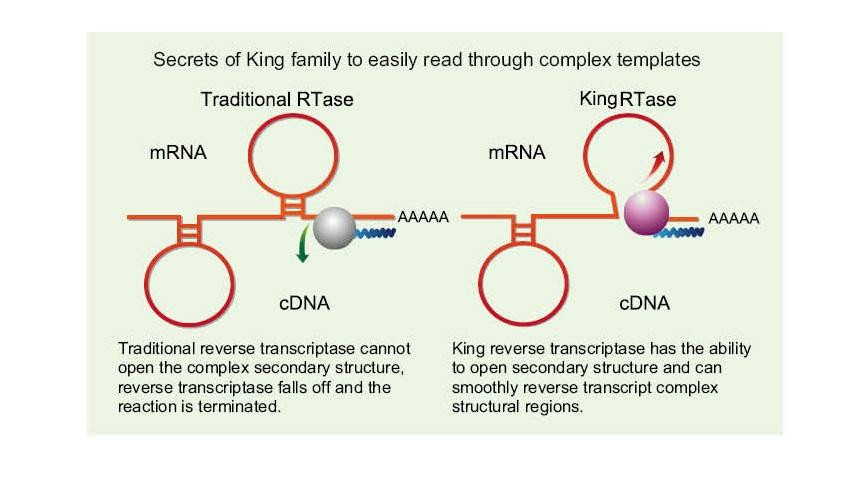
Leggi facilmente attraverso modelli complessi
——Leggi facilmente attraverso modelli complessi e GC elevati
L'RNA a filamento singolo ha una vasta gamma di regioni complesse della struttura secondaria a causa del legame idrogeno tra i filamenti. La normale trascrittasi inversa può portare alla terminazione della trascrizione inversa quando si incontra una struttura secondaria complessa, quindi incapace di completare con successo la sintesi del cDNA. Tuttavia, la nuova generazione di trascrittasi inversa King ha un dominio strutturale unico, che può distruggere il legame idrogeno tra i filamenti di RNA, aprendo così la complessa struttura secondaria dell'RNA e garantendo la trascrizione inversa regolare.
Tutti i prodotti possono essere personalizzati per ODM/OEM. Per dettagli,fare clic su Servizio personalizzato (ODM/OEM)
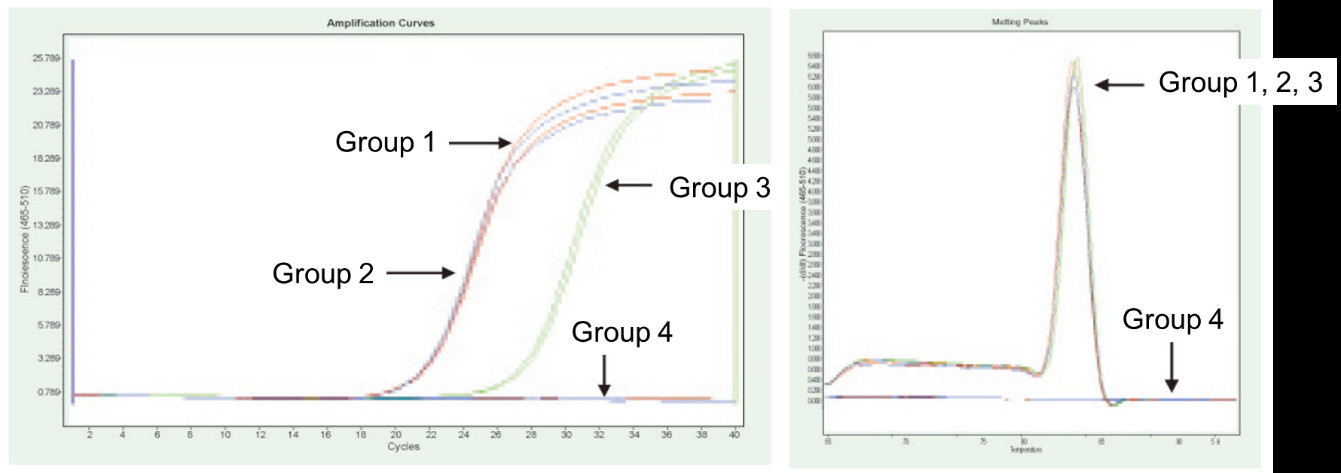 |
Gruppo 1: Trascrizione inversa senza trattamento con gDNasi; Gruppo 2: nessun trattamento con gDNasi e nessuna trascrizione inversa; Gruppo 3: trascrizione inversa dopo trattamento con gDNasi; Gruppo 4: trattamento con gDNasi senza trascrizione inversa. Metodi: Rilevazione PCR quantitativa a fluorescenza del gene TNF-alfa (primer progettato sull'esone con cDNA o genoma come modello) utilizzando 1 μg di RNA di cellule Hela (con residuo genomico) come modello. Risultati: come mostrato nella figura, il gruppo 2 può riflettere il residuo del genoma nell'RNA, il gruppo 3 può riflettere accuratamente il vero livello di espressione di TNF-alfa, il gruppo 1 ha errori nei risultati quantitativi finali dovuti al residuo del genoma e il gruppo 4 mostra che il kit FastKing RT può rimuovere completamente il DNA genomico residuo in RNA. |
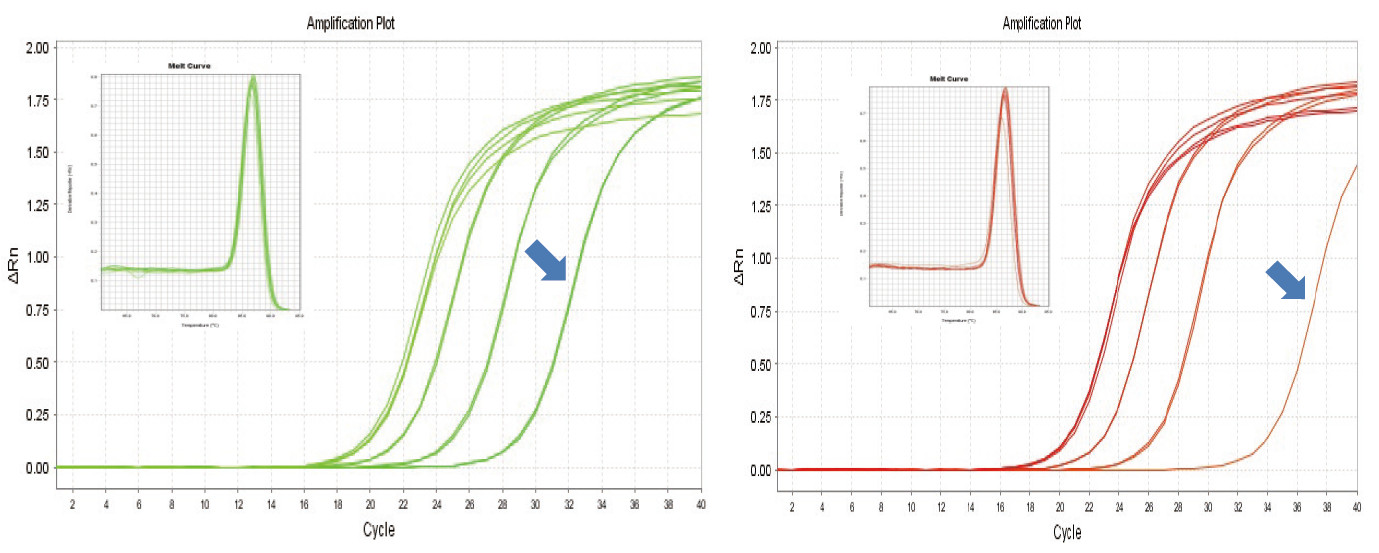 |
Figura 1. La trascrizione inversa dell'RNA di topo è stata eseguita utilizzando il kit TIANGEN FastKing RT (a sinistra) e il prodotto pertinente del fornitore A (a destra), quindi il gene MM5 è stato amplificato quantitativamente utilizzando TIANGEN SuperReal PreMix Plus (SYBR Green). Sono state analizzate la curva di amplificazione e la curva di fusione. L'input di RNA era rispettivamente di 1000 ng, 100 ng, 10 ng e 1 ng. I risultati mostrano che TIANGEN FastKing RT Kit ha un chiaro gradiente di trascrizione inversa e un basso valore Ct e presenta evidenti vantaggi per la trascrizione inversa del modello a bassa abbondanza (1 ng, freccia blu). |
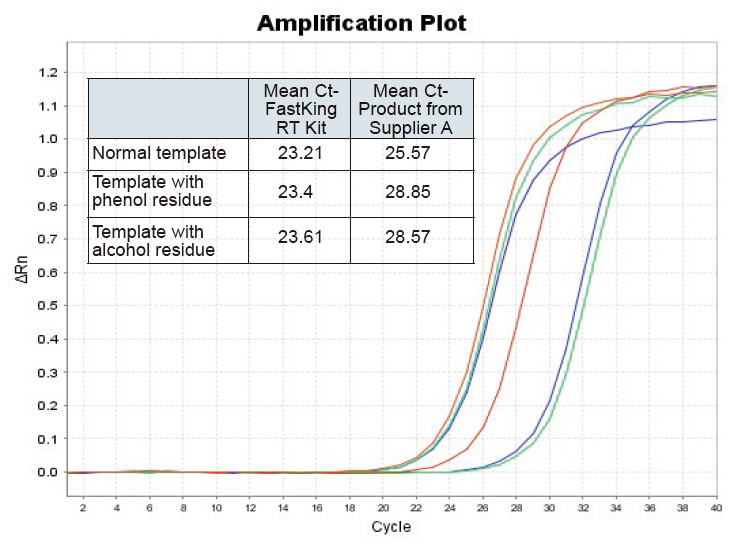 |
Figura 2. Trascrizione inversa del modello di RNA normale (rosso), modello con grande residuo fenolico (verde) e modello con residuo alcolico (blu) di ratti utilizzando rispettivamente il kit TIANGEN FastKing RT e il prodotto pertinente del fornitore A, quantificare i geni RNC utilizzando TIANGEN SuperReal Sono stati analizzati PreMix Plus (SYBR Green) e le curve di amplificazione e i valori Ct. I risultati mostrano che il kit TIANGEN FastKing RT ha il valore Ct quantitativo più basso dopo la trascrizione inversa e un'eccellente resistenza allo stress e presenta evidenti vantaggi per i modelli con residui di impurità elevate |
L'RNA A-1 è degradato
——Purifica RNA di alta qualità senza contaminazioni. Il materiale da cui viene estratto l'RNA dovrebbe essere il più fresco possibile per prevenire la degradazione dell'RNA. Analizzare l'integrità dell'RNA su gel denaturato prima della reazione RT. Dopo l'estrazione dell'RNA, dovrebbe essere conservato in formammide al 100%. Se viene utilizzato l'inibitore della RNasi, la temperatura di riscaldamento deve essere <45°C e il pH deve essere inferiore a 8,0, altrimenti l'inibitore rilascerà tutta la RNasi legata. Inoltre, l'inibitore della RNasi deve essere aggiunto in soluzioni contenenti ≥ 0,8 mM DTT.
L'RNA A-2 contiene inibitori delle reazioni di trascrizione inversa
——Gli inibitori della trascrizione inversa includono SDS, EDTA, glicerolo, pirofosfato di sodio, spermidina, formammide, sale di guanidina, ecc. Mescolare l'RNA di controllo con il campione e confrontare la resa con la reazione dell'RNA di controllo per verificare se è presente un inibitore. Lavare la precipitazione dell'RNA con etanolo al 70% (v/v) per rimuovere gli inibitori.
A-3 Ricottura insufficiente dei primer utilizzati per sintetizzare il primo filamento di cDNA
——Determinare che la temperatura di ricottura è adatta per i primer utilizzati nell'esperimento. Per esameri casuali, si consiglia di mantenere la temperatura a 25°C per 10 min prima di raggiungere la temperatura di reazione. Per i primer gene-specifici (GSP), prova altri GSP o passa a oligo(dT) o esamero casuale.
A-4 Piccola quantità di RNA di partenza
——Aumenta la quantità di RNA. Per campioni di RNA inferiori a 50 ng, è possibile utilizzare da 0,1 μg a 0,5 μg di acetil BSA nella sintesi del cDNA del primo filamento
A-5 La sequenza target non è espressa nei tessuti analizzati.
——Prova altri tessuti.
La reazione PCR A-6 fallisce
——Per la RT-PCR in due fasi, il modello di cDNA nella fase PCR non può superare 1/5 del volume di reazione.
A-1 Ricottura non specifica di primer e modelli
——L'estremità 3' dei primer non deve contenere 2-3 dG o dC. Utilizzare primer specifici del gene nella sintesi del primo filamento invece di primer casuali o oligo(dT). Utilizzare una temperatura di ricottura più alta nei primi cicli, quindi una temperatura di ricottura più bassa. Utilizzare la Taq DNA polimerasi hot-start per la PCR per migliorare la specificità della reazione.
A-2 Scarsa progettazione di primer gene-specifici
——Seguire gli stessi principi per la progettazione dei primer di amplificazione.
RNA A-3 contaminato con DNA genomico
——Trattare l'RNA con DNasi I di grado PCR. Impostare una reazione di controllo senza trascrizione inversa per rilevare la contaminazione del DNA.
A-4 Formazione di primer dimero
——Progettazione primer senza sequenze complementari all'estremità 3'.
A-5 Mg . troppo alto2+ concentrazione
——Ottimizza Mg2+ concentrazione per ogni combinazione stampo e primer
A-6 Contaminato con DNA estraneo
——Utilizzare punte resistenti agli aerosol ed enzimi UDG.
A-1 Il contenuto del prodotto del primo filamento è troppo alto
——Ridurre la quantità del prodotto del primo filamento nella fase di reazione PCR convenzionale.
A-2 Quantità di primer troppo elevata nella reazione PCR
——Riduce l'input del primer.
A-3 Troppi cicli
——Ottimizzare le condizioni di reazione PCR e ridurre il numero di cicli PCR.
A-4 Temperatura di ricottura troppo bassa
——Aumentare la temperatura di ricottura per prevenire l'inizio e l'estensione aspecifici.
A-5 Amplificazione non specifica di frammenti di oligonucleotidi generati dalla degradazione della DNasi del DNA —— Estrarre RNA di alta qualità per prevenire la contaminazione del DNA.
La RT-PCR consiste nel trascrivere inversamente l'RNA in cDNA e quindi utilizzare il cDNA trascritto inversamente come modello per la reazione di PCR per amplificare il frammento bersaglio. Scegli primer casuali, Oligo dT e primer specifici per gene in base alle condizioni specifiche dell'esperimento. Tutti i suddetti primer possono essere utilizzati per brevi mRNA di cellule eucariotiche senza struttura a forcina.
Primer casuale: adatto per RNA lungo con struttura a forcina, nonché tutti i tipi di RNA come rRNA, mRNA, tRNA, ecc. Sono utilizzati principalmente per la reazione RT-PCR di un singolo stampo.
Oligo dT: adatto per RNA con code PolyA (RNA procariotico, eucariotico Oligo dT rRNA e tRNA non hanno code PolyA). Poiché Oligo dT è legato alla coda PolyA, la qualità dei campioni di RNA deve essere elevata e anche una piccola quantità di degradazione ridurrà notevolmente la quantità di sintesi di cDNA a lunghezza intera.
Primer gene-specifico: complementare alla sequenza stampo, adatto a situazioni in cui la sequenza bersaglio è nota.
Ci sono due modi:
1. Metodo di riferimento interno: in teoria, il cDNA è costituito da frammenti di DNA di diverse lunghezze, quindi il risultato dell'elettroforesi è lo striscio. Se l'abbondanza di RNA è bassa, nessun prodotto verrà mostrato nell'elettroforesi, ma ciò non significa che nessun prodotto sarà amplificato dalla PCR. In generale, il riferimento interno può essere utilizzato per rilevare il cDNA. Se il riferimento interno ha risultati, la qualità del cDNA può essere sostanzialmente garantita (in alcuni casi, se il frammento del gene bersaglio è troppo lungo, possono esserci delle eccezioni).
2. Se esiste un gene noto amplificato da questo stampo, può essere verificato dai primer di questo gene. L'amplificazione del riferimento interno non significa necessariamente che non ci siano problemi con il cDNA. Poiché il riferimento interno ha un'elevata abbondanza nel cDNA, è facile da amplificare. Se il cDNA è parzialmente degradato per vari motivi, dal punto di vista della probabilità, i risultati della PCR di geni bersaglio a bassa abbondanza saranno notevolmente influenzati. Sebbene il riferimento interno sia ancora in abbondanza, l'amplificazione probabilmente non ne risentirà.
Degrado parziale dell'RNA. Rileva l'integrità e purifica l'RNA
Il contenuto di RNA di specie diverse può essere diverso, ma in generale, l'RNA totale estratto dovrebbe contenere due bande 28S e 18S chiare nell'elettroforesi su gel e la luminosità della prima banda dovrebbe essere il doppio di quella della seconda. La banda 5S indica che l'RNA è stato degradato e la sua luminosità è proporzionale al grado di degradazione. L'amplificazione riuscita del riferimento interno non significa che non ci siano problemi con l'RNA, poiché il riferimento interno è in alta abbondanza, l'RNA può essere amplificato purché la degradazione non sia grave. L'OD260/OD280il rapporto di RNA puro misurato dallo spettrofotometro dovrebbe essere compreso tra 1,9 e 2,1. Una piccola quantità di impurità proteiche nell'RNA ridurrà il rapporto. Finché il valore non è troppo basso, RT non sarà influenzato. Ciò che conta di più per RT è l'integrità dell'RNA.
L'estensione del gene di riferimento interno può solo indicare che la RT ha avuto successo, ma non è necessariamente correlata alla qualità del filamento di cDNA. Poiché i frammenti di riferimento interni sono generalmente di piccole dimensioni e ad alta espressione, è più facile avere successo nella trascrizione inversa. Tuttavia, la dimensione e l'espressione del gene bersaglio variano da gene a gene. La qualità del cDNA non può essere giudicata solo per riferimento interno, specialmente per i frammenti bersaglio più lunghi di 2 kb.
Alcuni campioni hanno strutture secondarie complesse, o hanno un ricco contenuto di GC, o sono preziosi con scarsa abbondanza. In questi casi, dovrebbe essere selezionata la trascrittasi inversa appropriata in base alle dimensioni del frammento bersaglio e del campione. Per modelli di RNA con alto contenuto di GC e struttura secondaria complessa, è difficile aprire la struttura secondaria a bassa temperatura o con la comune trascrittasi inversa. Per questi modelli, è possibile selezionare Quant Reverse Transcriptase, poiché le sue prestazioni di trascrizione inversa sono ovviamente migliori di quelle della trascrittasi inversa della serie M-MLV, che può invertire la trascrizione inversa di vari modelli di RNA in modo efficiente e trascrivere l'RNA nel primo filamento di cDNA nella misura massima. Quando si utilizza un kit di trascrittasi inversa generale, il sistema da 20 μl può solo trascrivere in modo inverso 1 μg di RNA totale. Si prega di prestare attenzione alla capacità massima RT del kit. Se il modello viene aggiunto in eccesso, la trascrizione inversa favorirà l'RNA con elevata abbondanza. Pertanto, è meglio non superare la capacità massima del sistema.
A-1 Determinare se l'RNA è gravemente degradato e se la RT ha successo
In generale, il motivo del fallimento dell'amplificazione del riferimento interno è spesso causato da una grave degradazione dell'RNA. Un altro possibile motivo è il fallimento della trascrizione inversa. Il riferimento interno non può essere utilizzato come standard per giudicare la qualità del singolo filamento di cDNA, ma può essere utilizzato come standard per giudicare se la trascrizione inversa ha successo se non ci sono problemi di qualità dell'RNA. La cosa più importante nel processo di trascrizione inversa è mantenere una temperatura costante e un sistema di reazione costante al fine di migliorare l'efficienza della reazione.
A-2 Determinare se i primer per l'amplificazione dei geni di riferimento interni sono affidabili e se ci sono problemi con i reagenti utilizzati nella PCR.
Per la quantificazione relativa, l'RNA deve essere quantificato prima della trascrizione inversa, che è richiesta anche in molti kit di trascrizione inversa, ad esempio quantificare l'input di RNA come 1 μg. Poiché il cDNA a trascrizione inversa è una soluzione mista, che include RNA, oligo dT, enzima, dNTP e persino un piccolo residuo di DNA, verrà causata una deviazione, quindi è impossibile quantificare con precisione il cDNA. Pertanto, è necessaria la quantificazione dell'RNA. Considerando che l'efficienza della trascrizione inversa è la stessa tra campioni diversi, la quantità di cDNA ottenuta dovrebbe essere la stessa e l'analisi quantitativa può mostrare il confronto dei livelli di espressione di diversi geni nella stessa quantità di RNA totale. Quando si esegue la PCR quantitativa a fluorescenza relativa, il cDNA quantitativo potrebbe non essere richiesto dopo la trascrizione inversa perché il gene di riferimento interno può fungere da riferimento.
È principalmente correlato ai geni e la trascrizione inversa del lungo frammento non è fattibile per la maggior parte dei geni. In primo luogo, l'efficienza della trascrizione inversa è di gran lunga inferiore a quella della PCR. In secondo luogo, la regione ricca di GC e la struttura secondaria di molti geni limitano sia la trascrizione inversa che la PCR. Infine, la fedeltà e l'efficienza di amplificazione della PCR sono difficili da garantire contemporaneamente. Nel processo di trascrizione inversa, nessuno può garantire di ottenere un lungo frammento per geni a bassa copia, specialmente usando oligo dT. Per quanto riguarda 5' UTR con più GC, è ancora più difficile. Pertanto, è ancora un metodo ragionevole per invertire la trascrizione con primer casuali, trovare i siti di scissione naturali nel frammento bersaglio, amplificare per segmenti e quindi eseguire la digestione di restrizione e la legatura. In generale è difficile amplificare direttamente frammenti più grandi di 2 kb, ma non sempre è impossibile ottenere: 1. Innanzitutto garantire l'integrità dell'RNA/mRNA, e si preferisce l'estrazione con TRIZOL. 2. Il kit M-MLV RT-PCR può essere utilizzato direttamente. Allungare il tempo di ricottura e aumentare correttamente il numero di cicli nel processo di amplificazione. In alternativa, è possibile applicare la PCR nidificata o eseguire prima una o due reazioni con una denaturazione e un tempo di estensione opportunamente estesi prima della normale amplificazione della PCR, che può aiutare ad estendere i frammenti. Prestare attenzione alla fedeltà della polimerasi. 3.Long Taq può essere utilizzato nella PCR per ottenere risultati ideali. 4.Per l'applicazione dell'espressione proteica, deve essere applicata la polimerasi ad alta fedeltà.
Ci sono due tipi di trascrittasi inversa offerti da TIANGEN: Quant/King RTase e TIANScript M-MLV. La principale differenza tra loro è la quantità di modelli di input. Quant è una trascrittasi inversa unica, diversa dall'M-MLV comunemente usato derivato dal virus della leucemia murina Moloney. Quant è una nuova trascrittasi inversa ad alta efficienza espressa in modo ricombinante dall'ingegneria Escherichia coli. Quant è adatto per amplificare 50 ng-2 μg di RNA con elevata attività trascrizionale inversa e alta resa. Rispetto al normale MMLV o AMV, la più grande caratteristica di Quant è che ha un'affinità molto forte con i modelli di RNA e può invertire i modelli complessi di trascrizione senza denaturazione ad alta temperatura. Per i modelli con un contenuto di GC più elevato, l'efficienza inversa è maggiore. Tuttavia, questa trascrittasi inversa ha attività RNasi H, che può influenzare la lunghezza del prodotto cDNA (adatto per modelli < 4,5 kb). Per la trascrizione inversa convenzionale, si consiglia la trascrittasi inversa TIANScript MMLV. Questa RTasi è un enzima modificato con attività RNasi H molto debole, che è adatta per la sintesi di cDNA lunga (> 5 kb).
La trascrizione inversa one-step e l'amplificazione PCR vengono completate nella stessa provetta senza aprire il coperchio della provetta tra la sintesi del cDNA e l'amplificazione, il che è utile per ridurre la contaminazione. Poiché tutti i campioni di cDNA ottenuti vengono utilizzati per l'amplificazione, la sensibilità è maggiore, con un minimo di 0,01 pg di RNA totale. Per un RTPCR one-step di successo, vengono generalmente utilizzati primer gene-specifici per avviare la sintesi del cDNA. Il metodo in due fasi, vale a dire la trascrizione inversa e l'amplificazione PCR, viene eseguito in due fasi. Innanzitutto viene eseguita la trascrizione inversa da uno stampo di RNA per ottenere il cDNA e il cDNA ottenuto viene sottoposto a una o più diverse reazioni di PCR. Il metodo in due fasi può utilizzare oligo(dT) o primer casuali per guidare la sintesi del primo filamento di cDNA e può invertire la trascrizione di tutte le informazioni sull'mRNA da un campione specifico.
Categorie di prodotti
PERCHÉ SCEGLIERE NOI
Dalla sua istituzione, la nostra fabbrica ha sviluppato prodotti di prima classe mondiale con l'adesione al principio
di qualità prima di tutto. I nostri prodotti hanno guadagnato un'eccellente reputazione nel settore e preziosa fiducia tra i clienti vecchi e nuovi.










