Mix di amplificazione HiFi TIANSeq
A valle
Amplificazione PCR della libreria NGS, amplificazione PCR con sequenziamento di prima generazione, clonazione ad alta fedeltà, rilevamento SNP, mutazione sito-specifica, ecc.
Caratteristiche
■ Amplificazione ad alta efficienza: garantisce il tasso di conversione e riduce i cicli di amplificazione.
■ Bassa preferenza: efficienza di amplificazione bilanciata per modelli di DNA con diversi contenuti di GC%.
■ Alta specificità: con proprietà HotStart e forte specificità.
■ Alta fedeltà: la fedeltà è 50 volte più alta della Taq DNA polimerasi.
■ Alta sensibilità: l'input del modello può essere a partire da 1 pag.
Tutti i prodotti possono essere personalizzati per ODM/OEM. Per dettagli,fare clic su Servizio personalizzato (ODM/OEM)
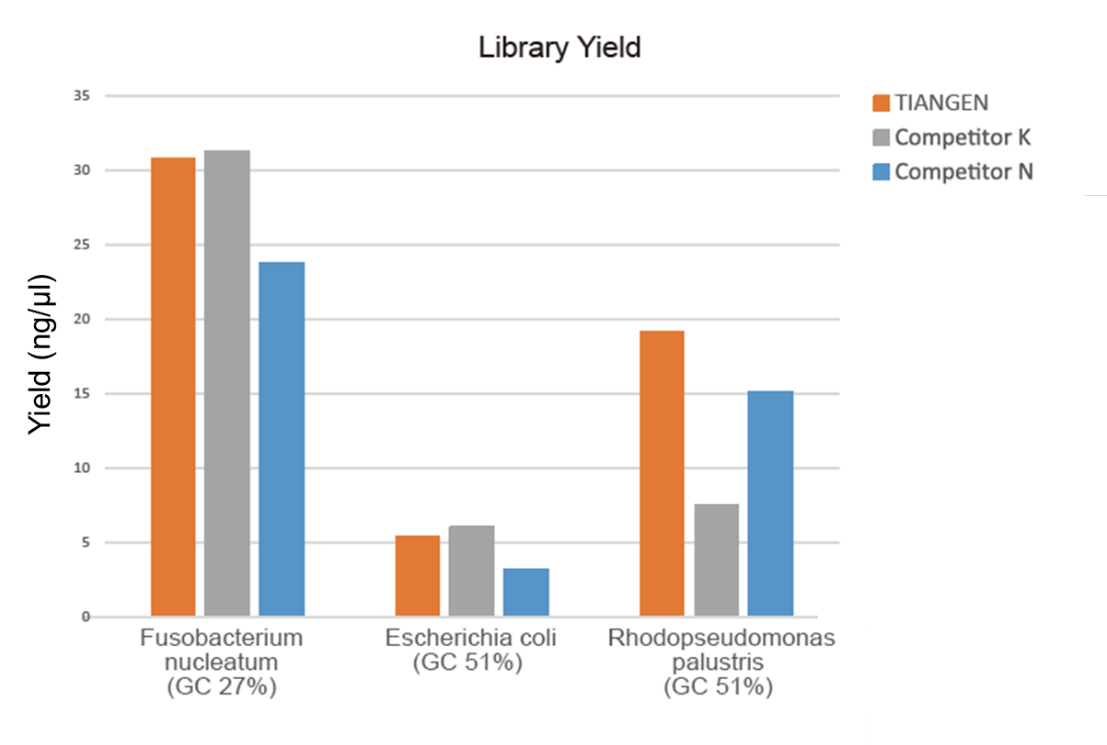 |
Figura 1. L'arricchimento della libreria del DNA genomico con diversi rapporti GC (input genomico 10 ng, amplificazione di 8 cicli) è stato effettuato simultaneamente utilizzando TIANSeq HiFi Amplification Mix e l'enzima HiFi del fornitore K e N e la resa della libreria è stata rilevata da Agilent 2100. I risultati hanno mostrato che TIANSeq HiFi Amplification Mix ha un'elevata resa della libreria, con una forte universalità per diversi contenuti GC, e le prestazioni di arricchimento della libreria sono state migliori rispetto ad altri fornitori. |
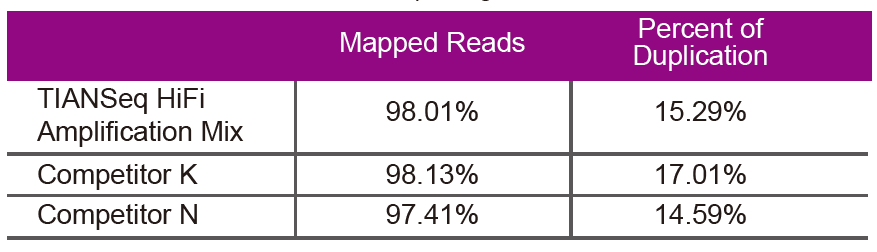 |
Dati di sequenziamentoUtilizzare la miscela di amplificazione HiFi TIANSeq e l'enzima HiFi appositamente utilizzati per l'amplificazione della libreria NGS dal fornitore K e N per l'amplificazione della libreria dello stesso DNA genomico (l'input del genoma è 10 ng). Dopo il sequenziamento, analizzare la copertura e il tasso di duplicazione della libreria. |
Preferenza GC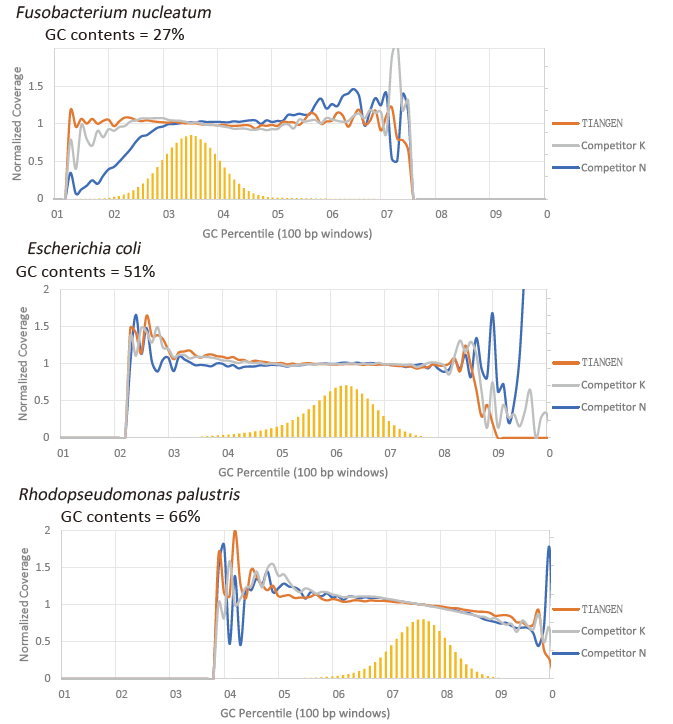 |
Figura 2. Amplificazione delle librerie di genomi con diversi contenuti CG utilizzando TIANSeq HiFi Amplification Mix e HIFi del fornitore K e N. Il risultato mostra che l'uniformità della libreria di amplificazione TIANSeq HiFi Amplification Mix è buona e senza preferenza GC, che è equivalente ai risultati dell'azienda K e leggermente migliore dei prodotti dell'azienda N. I risultati mostrano che la copertura della libreria di amplificazione TIANSeq HiFi Amplification Mix è elevata, il tasso di duplicazione ha soddisfatto i requisiti e le prestazioni di amplificazione della libreria sono equivalenti a quelle della concorrenza. |
Attualmente, la tecnologia di sequenziamento ad alto rendimento si basa principalmente sulla tecnologia di sequenziamento di nuova generazione. Poiché la lunghezza di lettura della tecnologia di sequenziamento di nuova generazione è limitata, dobbiamo suddividere la sequenza a lunghezza intera in piccole librerie di frammenti da sequenziare. In base alle esigenze dei diversi esperimenti di sequenziamento, di solito scegliamo il sequenziamento a terminazione singola o il sequenziamento a doppia estremità. Attualmente i frammenti di DNA della libreria di sequenziamento di nuova generazione sono generalmente distribuiti nell'intervallo 200-800 bp.

a) Il DNA è di scarsa qualità e contiene inibitori. Utilizzare campioni di DNA di alta qualità per evitare l'inibizione dell'attività enzimatica.
b) La quantità di campione di DNA è insufficiente quando si utilizza il metodo PCR-free per costruire la libreria del DNA. Quando l'input del DNA frammentato supera i 50 ng, il flusso di lavoro privo di PCR può essere eseguito in modo selettivo durante il processo di costruzione della libreria. Se il numero di copie della libreria è troppo basso per essere sequenziato direttamente, la libreria del DNA può essere amplificata mediante PCR dopo la legatura dell'adattatore.
c) La contaminazione da RNA porta a una quantificazione iniziale del DNA imprecisa La contaminazione da RNA può esistere nel processo di purificazione del DNA genomico, che può portare a una quantificazione imprecisa del DNA ea un caricamento insufficiente del DNA durante la costruzione della libreria. L'RNA può essere rimosso trattando con RNasi.
LA-1
a) Compaiono piccoli frammenti (60 bp-120 bp) I piccoli frammenti sono solitamente frammenti di adattatori o dimeri formati da adattatori. La purificazione con le sfere magnetiche Agencourt AMPure XP può rimuovere efficacemente questi frammenti dell'adattatore e garantire la qualità del sequenziamento.
b) Frammenti di grandi dimensioni compaiono nella libreria dopo l'amplificazione PCR La dimensione del frammento di DNA della libreria aumenterà di 120 bp dopo la legatura dell'adattatore. Se il frammento di DNA aumenta di oltre 120 bp dopo la legatura dell'adattatore, potrebbe essere causato da un'amplificazione anormale del frammento di un'eccessiva amplificazione PCR. Ridurre il numero di cicli PCR può prevenire la situazione.
c) Dimensione anormale dei frammenti di DNA della libreria dopo la legatura dell'adattatore La lunghezza dell'adattatore in questo kit è di 60 bp. Quando le due estremità del frammento sono legate agli adattatori, la lunghezza aumenterà solo di 120 bp. Quando si utilizza un adattatore diverso da quello fornito da questo kit, contattare il fornitore per fornire informazioni rilevanti come la lunghezza dell'adattatore. Assicurati che il flusso di lavoro e l'operazione dell'esperimento seguano i passaggi descritti nel manuale.
d) Dimensione anormale del frammento di DNA prima della legatura dell'adattatore Il motivo di questo problema può essere causato da condizioni di reazione errate durante la frammentazione del DNA. Tempi di reazione diversi dovrebbero essere usati per diversi input di DNA. Se l'input del DNA è superiore a 10 ng, si consiglia di scegliere il tempo di reazione di 12 min come tempo di inizio per l'ottimizzazione e la dimensione del frammento prodotta in questo momento è principalmente nell'intervallo 300-500 bp. Gli utenti possono aumentare o diminuire la lunghezza dei frammenti di DNA per 2-4 minuti in base alle proprie esigenze per ottimizzare i frammenti di DNA con la dimensione richiesta.
A-2
a) Il tempo di frammentazione non è ottimizzato Se il DNA frammentato è troppo piccolo o troppo grande, fare riferimento alle Linee guida per la selezione del tempo di frammentazione fornite nelle istruzioni per determinare il tempo di reazione e utilizzare questo punto temporale come controllo, inoltre impostare un sistema di reazione per prolungare o accorciare 3 min per effettuare una regolazione più accurata sul tempo di frammentazione.
LA-3
Distribuzione anormale delle dimensioni del DNA dopo il trattamento di frammentazione
a) Metodo di scongelamento errato del reagente di frammentazione o il reagente non è completamente miscelato dopo lo scongelamento. Scongelare il reagente 5× Fragmentation Enzyme Mix su ghiaccio. Una volta scongelato, mescolare uniformemente il reagente scuotendo delicatamente il fondo della provetta. Non agitare il reagente!
b) Il campione di DNA in ingresso contiene EDTA o altri inquinanti L'esaurimento degli ioni sale e degli agenti chelanti nella fase di purificazione del DNA è particolarmente importante per il successo dell'esperimento. Se il DNA viene sciolto in 1×TE, utilizzare il metodo fornito nelle istruzioni per eseguire la frammentazione. Se la concentrazione di EDTA nella soluzione è incerta, si consiglia di purificare il DNA e dissolverlo in acqua deionizzata per la successiva reazione.
c) Quantificazione iniziale del DNA imprecisa La dimensione del DNA frammentato è strettamente correlata alla quantità di DNA in ingresso. Prima del trattamento di frammentazione, è essenziale una quantificazione accurata del DNA utilizzando Qubit, Picogreen e altri metodi per determinare l'esatta quantità di DNA nel sistema di reazione.
d) La preparazione del sistema di reazione non segue le istruzioni La preparazione del sistema di reazione frammentato deve essere eseguita su ghiaccio rigorosamente secondo le istruzioni. Per garantire l'effetto migliore, tutti i componenti della reazione devono essere posti su ghiaccio e la preparazione del sistema di reazione deve essere eseguita dopo il completo raffreddamento. Una volta completata la preparazione, agitare o pipettare per mescolare accuratamente. Non vortice!
1. Un metodo di miscelazione improprio (vortice, oscillazione violenta, ecc.) causerà una distribuzione anomala dei frammenti della libreria (come mostrato nella figura seguente), influenzando così la qualità della libreria. Pertanto, quando si prepara la soluzione di reazione della miscela di frammentazione, pipettare delicatamente su e giù per miscelare o utilizzare la punta del dito per scuotere e miscelare in modo uniforme. Fare attenzione a non mescolare con il vortice.

2. Per la costruzione della biblioteca deve essere utilizzato DNA ad elevata purezza
■ Buona integrità del DNA: la banda dell'elettroforesi è superiore a 30 kb, senza code
■ OD260/230: >1,5
■ OD260/280: 1,7-1,9
3. La quantità di DNA immessa deve essere accurata Si suggerisce di utilizzare i metodi Qubit e PicoGreen per quantificare il DNA, piuttosto che Nanodrop.
4. Il contenuto di EDTA nella soluzione di DNA deve essere determinato L'EDTA ha una grande influenza sulla reazione di frammentazione. Se il contenuto di EDTA è elevato, è necessario eseguire la purificazione del DNA prima del test successivo.
5. La soluzione di reazione di frammentazione deve essere preparata su ghiaccio. Il processo di frammentazione è sensibile alla temperatura e al tempo di reazione (soprattutto dopo l'aggiunta di potenziatore). Per garantire l'accuratezza del tempo di reazione, preparare il sistema di reazione su ghiaccio.
6. Il tempo di reazione alla frammentazione deve essere accurato Il tempo di reazione della fase di frammentazione influenzerà direttamente la dimensione dei prodotti del frammento, influenzando così la distribuzione delle dimensioni dei frammenti di DNA nella libreria.
1. Che tipo di campione è applicabile a questo kit?
Il tipo di campione applicabile di questo kit può essere RNA totale o mRNA purificato con una buona integrità dell'RNA. Se viene utilizzato l'RNA totale per costruire la libreria, si consiglia di utilizzare il kit di deplezione dell'rRNA (Cat#4992363/4992364/4992391) per rimuovere prima l'rRNA.
2. È possibile utilizzare campioni FFPE per creare una libreria con questo kit?
L'mRNA nei campioni FFPE sarà degradato in una certa misura, con relativa scarsa integrità. Quando si utilizza questo kit per la costruzione della libreria, si consiglia di ottimizzare il tempo di frammentazione (abbreviare il tempo di frammentazione o non eseguire la frammentazione).
3. Utilizzando il passaggio di selezione delle dimensioni fornito nel manuale del prodotto, cosa potrebbe causare una leggera deviazione del segmento inserito?
La selezione della taglia deve essere eseguita in stretta conformità con la fase di selezione della taglia in questo manuale del prodotto. Se c'è una deviazione, il motivo potrebbe essere che le sfere magnetiche non sono bilanciate a temperatura ambiente o non sono completamente miscelate, la pipetta non è precisa o il liquido è rimasto nel puntale. Si consiglia di utilizzare i puntali a basso assorbimento per l'esperimento.
4. Selezione degli adattatori nella costruzione delle biblioteche
Il kit di costruzione della libreria non contiene il reagente dell'adattatore e si consiglia di utilizzare questo kit insieme all'adattatore TIANSeq a indice singolo (Illumina) (4992641/492642/49923).
5. Controllo di qualità della biblioteca
Rilevamento quantitativo della libreria: Qubit e qPCR vengono utilizzati per determinare rispettivamente la concentrazione di massa e la concentrazione molare della libreria. L'operazione è rigorosamente conforme al manuale del prodotto. La concentrazione della libreria generalmente soddisferà i requisiti del sequenziamento NGS. Rilevamento dell'intervallo di distribuzione della libreria: utilizzo di Agilent 2100 Bioanalyzer per rilevare l'intervallo di distribuzione della libreria.
6. Selezione del numero del ciclo di amplificazione
Secondo le istruzioni, il numero di cicli PCR è 6-12 e il numero di cicli PCR necessari deve essere selezionato in base all'input del campione. Nelle librerie ad alto rendimento, di solito si verifica un'amplificazione eccessiva in vari gradi, che si manifesta con un picco leggermente più grande dopo il picco dell'intervallo target nel rilevamento di Agilent 2100 Bioanalyzer, oppure la concentrazione rilevata di Qubit è inferiore a quella di qPCR. L'amplificazione lievemente eccessiva è un fenomeno normale, che non influisce sul sequenziamento della libreria e sulla successiva analisi dei dati.
7. I picchi vengono visualizzati nel profilo di rilevamento di Agilent 2100 Bioanalyzer
La comparsa di picchi nella rilevazione del bioanalizzatore Agilent 2100 è dovuta alla frammentazione non uniforme dei campioni, dove ci saranno più frammenti di una certa dimensione, e questo diventerà più evidente dopo l'arricchimento tramite PCR. In questo caso, si suggerisce di non effettuare la selezione dimensionale, cioè impostare la condizione di frammentazione a 94°C per 15 min incubati, dove la distribuzione del frammento è piccola e concentrata e l'omogeneità può essere migliorata.
Categorie di prodotti
PERCHÉ SCEGLIERE NOI
Dalla sua istituzione, la nostra fabbrica ha sviluppato prodotti di prima classe mondiale con l'adesione al principio
di qualità prima di tutto. I nostri prodotti hanno guadagnato un'eccellente reputazione nel settore e preziosa fiducia tra i clienti vecchi e nuovi.








