TIANquick FFPE DNA Kit
Caratteristiche
■ L'intera procedura potrebbe essere completata entro un'ora.
■ Non sono necessari né xilene né altri solventi pericolosi.
■ Il DNA di elevata purezza può essere isolato senza digestione notturna della proteinasi K.
Applicazioni
■ PCR e RT-qPCR
■ Analisi dei geni SNP e STR
■ Ricerca farmacogenomica
Tutti i prodotti possono essere personalizzati per ODM/OEM. Per dettagli,fare clic su Servizio personalizzato (ODM/OEM)
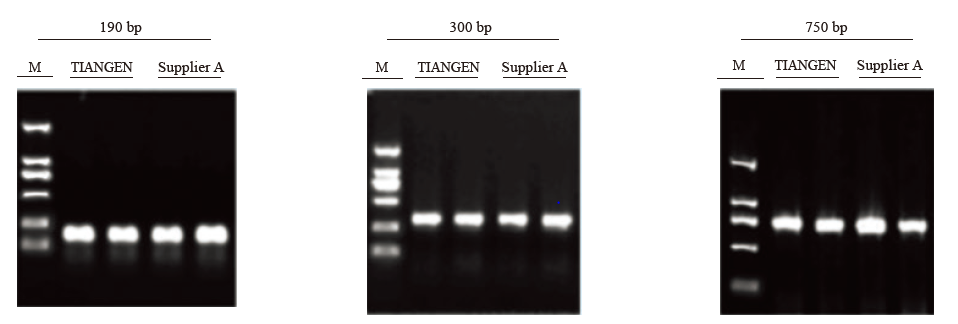
Il DNA genomico estratto dal kit TIANquick FFPE DNA e il relativo prodotto dal fornitore A sono stati amplificati a frammenti di 190 bp, 300 bp, 750 bp. Il risultato mostra che le efficienze di amplificazione PCR del gDNA estratto dal kit TIANquick FFPE DNA e dal fornitore A erano equivalenti.
Materiale di partenza: campioni FFPE di fegato di topo (5 pezzi, 10 μm/pezzo)
Procedura: seguire le istruzioni di ciascun prodotto.
Caricamento del gel:
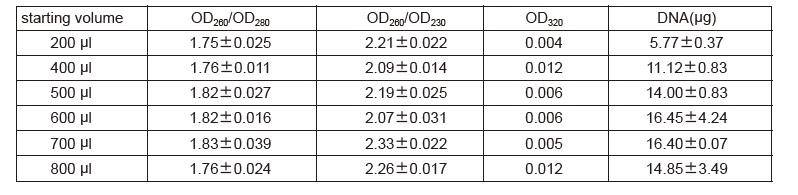
Il gDNA è stato eluito con 50 μl di tampone di eluizione ed è stato amplificato a 190 bp, 300 bp e 750 bp. 5 μl di 20 μl di prodotti PCR sono stati caricati per corsia su gel di agarosio all'1%. L'elettroforesi è stata condotta a 6 V/cm per 20 min.
M: Marcatore TIANGEN D2000;
T: kit DNA TIANquick FFPE;
A: Prodotto simile dal fornitore A.
A-1 Bassa concentrazione di cellule o virus nel campione di partenza —Arricchire la concentrazione di cellule o virus.
A-2 Lisi dei campioni insufficiente —I campioni non sono stati miscelati accuratamente con il tampone di lisi. Si consiglia di miscelare accuratamente con vortex a impulsi per 1-2 volte. —Lisi cellulare insufficiente causata dalla diminuzione dell'attività della proteinasi K. —Lisi cellulare o degradazione delle proteine insufficiente a causa del tempo insufficiente del bagno caldo. Si consiglia di tagliare il tessuto in piccoli pezzi e prolungare il tempo del bagno per rimuovere tutti i residui nel lisato.
A-3 Adsorbimento di DNA insufficiente. —Nessun etanolo o una bassa percentuale invece del 100% di etanolo è stato aggiunto prima che il lisato fosse trasferito alla colonna spin.
A-4 Il valore del pH del tampone di eluizione è troppo basso. —Regolare il pH tra 8,0-8,3.
Etanolo residuo nell'eluente.
—Vi è residuo di tampone di lavaggio PW nell'eluente. L'etanolo può essere rimosso centrifugando la colonna spin per 3-5 min, e poi ponendola a temperatura ambiente o in un incubatore a 50 per 1-2 min.
A-1 Il campione non è fresco. —Estrarre un campione di DNA positivo come controllo per determinare se il DNA nel campione si è degradato.
A-2 Pretrattamento improprio. —Causa da un'eccessiva macinazione dell'azoto liquido, dalla ripresa di umidità o da una quantità eccessiva di campione.
I pretrattamenti dovrebbero variare per i diversi campioni. Per i campioni di piante, assicurati di macinare accuratamente in azoto liquido. Per i campioni animali, omogeneizzare o macinare accuratamente in azoto liquido. Per campioni con pareti cellulari difficili da rompere, come batteri G+ e lieviti, si consiglia di utilizzare lisozima, liticasi o metodi meccanici per rompere le pareti cellulari.
4992201/4992202 Plant Genomic DNA Kit adotta un metodo basato su colonne che richiede cloroformio per l'estrazione. È particolarmente adatto per vari campioni di piante, nonché per la polvere secca di piante. Anche Hi-DNAsecure Plant Kit è a base di colonne, ma non necessita di estrazione con fenolo/cloroformio, il che lo rende sicuro e non tossico. È adatto per piante con alto contenuto di polisaccaridi e polifenoli. 4992709/4992710 DNAquick Plant System adotta un metodo a base liquida. Anche l'estrazione di fenolo/cloroformio non è necessaria. La procedura di purificazione è semplice e veloce senza limiti per le quantità iniziali del campione, in modo che gli utenti possano regolare la quantità in modo flessibile in base ai requisiti sperimentali. Grandi dimensioni di frammenti di gDNA possono essere ottenute con un'elevata resa.
L'estrazione del DNA del coagulo di sangue può essere eseguita utilizzando i reagenti forniti in questi due kit semplicemente modificando il protocollo con le istruzioni specifiche per l'estrazione del DNA del coagulo di sangue. La copia elettronica del protocollo di estrazione del DNA del coagulo di sangue può essere rilasciata su richiesta.
Sospendere il campione fresco con 1 ml di PBS, soluzione salina normale o tampone TE. Omogeneizzare completamente il campione con un omogeneizzatore e raccogliere il precipitato sul fondo di una provetta mediante centrifugazione. Eliminare il surnatante e risospendere il precipitato con 200 microlitri di tampone GA. La seguente purificazione del DNA può essere eseguita secondo le istruzioni.
Per la purificazione del gDNA in campioni di plasma, siero e fluidi corporei, si consiglia il kit TIANamp Micro DNA. Per la purificazione del gDNA del virus da campioni di siero/plasma, si consiglia il kit TIANamp Virus DNA/RNA. Per la purificazione del gDNA batterico da campioni di siero e plasma, si consiglia il kit TIANamp Bacteria DNA (il lisozima deve essere incluso per i batteri positivi). Per i campioni di saliva, sono consigliati il kit Hi-Swab DNA e il kit TIANamp Bacteria DNA.
DNAsecure Plant Kit o DNAquick Plant System sono raccomandati per l'estrazione del genoma fungino. Per l'estrazione del genoma del lievito, si consiglia il kit TIANamp Yeast DNA (la liticasi deve essere preparata autonomamente).
Categorie di prodotti
PERCHÉ SCEGLIERE NOI
Dalla sua istituzione, la nostra fabbrica ha sviluppato prodotti di prima classe mondiale con l'adesione al principio
di qualità prima di tutto. I nostri prodotti hanno guadagnato un'eccellente reputazione nel settore e preziosa fiducia tra i clienti vecchi e nuovi.