Kit per PCR diretto del sangue
Caratteristiche
■ Semplice e veloce: l'amplificazione PCR può essere eseguita direttamente utilizzando il sangue come stampo, senza la necessità delle noiose fasi della preparazione del campione e dell'estrazione del DNA.
■ Elevata purezza: il mancato rispetto delle fasi di pretrattamento del campione e di estrazione del DNA può aiutare a evitare la contaminazione incrociata dei campioni.
■ Elevata produttività: l'identificazione della PCR per campioni su larga scala può essere eseguita combinando il kit con piastre PCR da 96/384 pozzetti.
■ Forte universalità: questo kit può amplificare in modo efficiente frammenti ad alto contenuto di GC o frammenti con struttura secondaria complessa e la lunghezza di amplificazione può arrivare fino a 5 kb.
■ Forte resistenza allo stress: questo kit può essere applicato a varie specie e campioni di sangue conservati in modi diversi.
Applicazioni
I prodotti PCR di questo kit contengono "A" all'estremità 3′, che può essere utilizzata direttamente per la clonazione del vettore TA. Questo kit può essere utilizzato per l'amplificazione di frammenti di DNA genomico, analisi genetiche ad alto rendimento e analisi di genotipizzazione (come il rilevamento di geni).
Tutti i prodotti possono essere personalizzati per ODM/OEM. Per dettagli,fare clic su Servizio personalizzato (ODM/OEM)
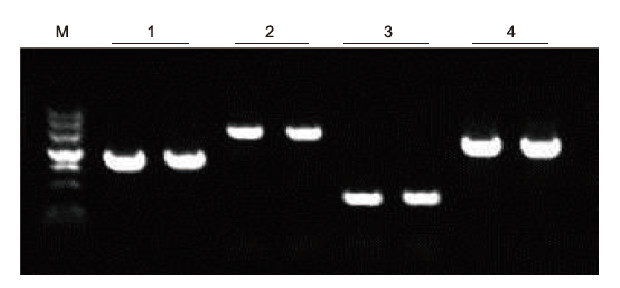 |
Usando l'anticoagulazione umana con EDTA come stampo, 4 geni con diversi contenuti di GC sono stati amplificati dal kit Blood Direct PCR. Il sistema di reazione PCR era di 20 μl e come stampo è stato utilizzato 1 μl di sangue. M: TIANGEN Marker II; 1: dimensione del frammento 1090 bp, contenuto GC 68,1%; 2: dimensione del frammento 1915 bp, contenuto GC 70,4%; 3: dimensione del frammento 448 bp, contenuto GC 74,8%; 4: Dimensione del frammento 1527 bp, contenuto GC 61,5%. Risultati sperimentali: il kit Blood Direct PCR può amplificare efficacemente i frammenti di DNA con il contenuto di GC nell'intervallo 61,5%-74,8%, suggerendo che è in grado di amplificare frammenti ad alto contenuto di GC. |
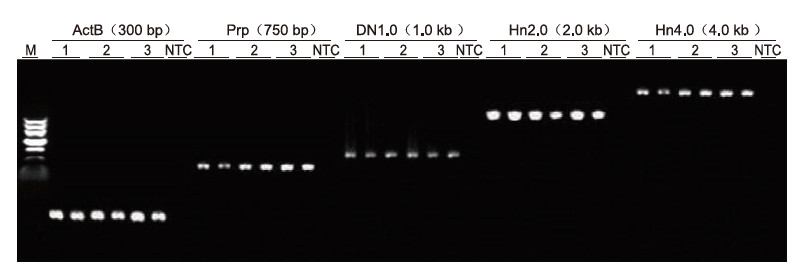 |
Utilizzando l'anticoagulazione EDTA umana come modello, 5 geni con lunghezze diverse (ActB, Prp, DN1.0, Hn2.0 e Hn4.0) sono stati amplificati dal kit Blood Direct PCR. Il sistema di reazione PCR era di 20 μl e come stampo è stato utilizzato 1 μl di sangue. M: TIANGEN Marker II; 1-3: 3 diversi campioni di sangue; NTC: controllo senza primer. Risultati sperimentali: il kit Blood Direct PCR può amplificare frammenti di lunghezza fino a 4 kb, suggerendo che è in grado di amplificare frammenti lunghi. |
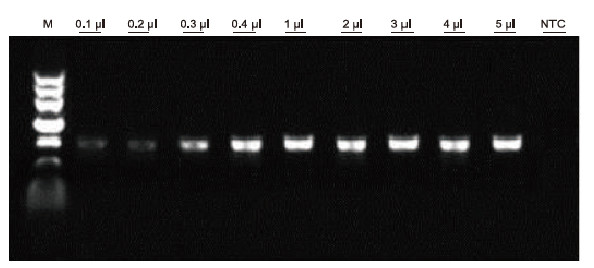 |
Utilizzando l'anticoagulante EDTA umano come modello, il kit Blood Direct PCR è stato utilizzato per il rilevamento tramite PCR di diversi campioni di sangue. Il sistema di reazione PCR era di 20 μl e come stampo è stato utilizzato 1 μl di sangue. M: TIANGEN Marker II; 1-9: la quantità di sangue caricata è 0,1 μl, 0,2 μl, 0,3 μl, 0,4 μl, 1 μl, 2 μl, 3 μl, 4 μl e 5 μl, rispettivamente; NTC: controllo senza template Risultati sperimentali: il kit Blood Direct PCR ha una forte resistenza al sangue e può amplificare i campioni di sangue con un intervallo di carico di 0,1-5 μl. |
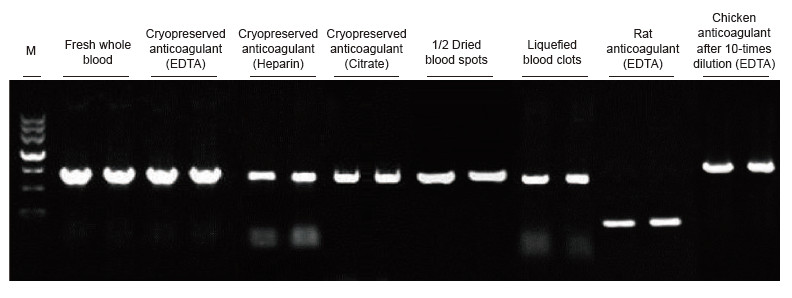 |
Come modelli sono stati utilizzati campioni di sangue di umani, ratti, polli e altre specie con trattamenti diversi. Il kit Blood Direct PCR è stato utilizzato per amplificare PRNP (umano, 750 bp), actina (ratto, 200 bp) e β-actina (pollo, 1,0 kb). Il sistema di reazione PCR era di 20 μl e come stampo è stato utilizzato 1 μl di sangue. M: TIANGEN Marker II. Risultati sperimentali: il kit Blood Direct PCR può essere applicato su un'ampia gamma di campioni e la rilevazione diretta tramite PCR può essere eseguita su campioni di sangue di varie specie con trattamenti diversi. |
Modello A-1
■ Il modello contiene impurità proteiche o inibitori Taq, ecc. ——Purificare il modello di DNA, rimuovere le impurità proteiche o estrarre il DNA modello con i kit di purificazione.
■ La denaturazione del templato non è completa ——Aumentare opportunamente la temperatura di denaturazione e prolungare il tempo di denaturazione.
■ Degrado del modello ——Ripreparare il modello.
A-2 Primer
■ Scarsa qualità dei primer ——Risintetizzare il primer.
■ Degradazione del primer ——Aliquotare i primer ad alta concentrazione in un piccolo volume per la conservazione. Evitare il congelamento e lo scongelamento multiplo o la crioconservazione a lungo termine a 4°C.
■ Progettazione inadeguata dei primer (es. lunghezza del primer non sufficiente, dimero formato tra i primer, ecc.) - Riprogettazione dei primer (evitare la formazione di dimero di primer e struttura secondaria)
A-3 mg2+concentrazione
■ Mg2+ la concentrazione è troppo bassa ——Aumentare adeguatamente Mg2+ concentrazione: Ottimizza il Mg2+ concentrazione mediante una serie di reazioni da 1 mM a 3 mM con un intervallo di 0,5 mM per determinare il Mg . ottimale2+ concentrazione per ogni stampo e primer.
A-4 Temperatura di ricottura
■ L'elevata temperatura di ricottura influisce sul legame del primer e del modello. ——Ridurre la temperatura di ricottura e ottimizzare la condizione con un gradiente di 2°C.
A-5 Tempo di estensione
■ Tempo di estensione breve: aumenta il tempo di estensione.
Fenomeni: i campioni negativi mostrano anche le bande della sequenza bersaglio.
A-1 Contaminazione della PCR
■ Contaminazione incrociata della sequenza target o dei prodotti di amplificazione ——Fare attenzione a non pipettare il campione contenente la sequenza target nel campione negativo oa versarli fuori dalla provetta da centrifuga. I reagenti o l'attrezzatura devono essere sterilizzati in autoclave per eliminare gli acidi nucleici esistenti e l'esistenza di contaminazione deve essere determinata mediante esperimenti di controllo negativo.
■ Contaminazione dei reagenti ——Aliquotare i reagenti e conservarli a bassa temperatura.
A-2 Primor
■ Mg2+ la concentrazione è troppo bassa ——Aumentare adeguatamente Mg2+ concentrazione: Ottimizza il Mg2+ concentrazione mediante una serie di reazioni da 1 mM a 3 mM con un intervallo di 0,5 mM per determinare il Mg . ottimale2+ concentrazione per ogni stampo e primer.
■ Progettazione del primer non corretta e la sequenza target ha omologia con la sequenza non target. ——Riprogettazione dei primer.
Fenomeni: le bande di amplificazione della PCR non sono coerenti con le dimensioni previste, sia grandi che piccole, o talvolta si verificano sia bande di amplificazione specifiche che bande di amplificazione non specifiche.
A-1 Primer
■ Scarsa specificità del primer
——Riprogettazione del primer.
■ La concentrazione di primer è troppo alta ——Aumentare adeguatamente la temperatura di denaturazione e prolungare il tempo di denaturazione.
A-2 mg2+ concentrazione
■ Il Mg2+ la concentrazione è troppo alta ——Ridurre adeguatamente la concentrazione di Mg2+: ottimizzare il Mg2+ concentrazione mediante una serie di reazioni da 1 mM a 3 mM con un intervallo di 0,5 mM per determinare il Mg . ottimale2+ concentrazione per ogni stampo e primer.
A-3 Polimerasi termostabile
■ Quantità eccessiva di enzima ——Ridurre la quantità di enzima in modo appropriato a intervalli di 0,5 U.
A-4 Temperatura di ricottura
■ La temperatura di ricottura è troppo bassa ——Aumentare opportunamente la temperatura di ricottura o adottare il metodo di ricottura a due fasi
A-5 cicli PCR
■ Troppi cicli PCR ——Ridurre il numero di cicli PCR.
A-1 Primer——Scarsa specificità ——Riprogettare il primer, modificare la posizione e la lunghezza del primer per migliorarne la specificità; o eseguire la PCR nidificata.
A-2 DNA modello
——Il modello non è puro ——Purificare il modello o estrarre il DNA con i kit di purificazione.
A-3 mg2+ concentrazione
——Mg2+ la concentrazione è troppo alta ——Ridurre adeguatamente Mg2+ concentrazione: Ottimizza il Mg2+ concentrazione mediante una serie di reazioni da 1 mM a 3 mM con un intervallo di 0,5 mM per determinare il Mg . ottimale2+ concentrazione per ogni stampo e primer.
A-4 dNTP
——La concentrazione di dNTP è troppo alta ——Ridurre adeguatamente la concentrazione di dNTP
A-5 Temperatura di ricottura
——Temperatura di ricottura troppo bassa ——Aumentare opportunamente la temperatura di ricottura
A-6 Cicli
——Troppi cicli ——Ottimizza il numero di cicli

Il primo passo è scegliere la polimerasi appropriata. La normale Taq polimerasi non può correggere le bozze a causa della mancanza di attività esonucleasica 3'-5' e la mancata corrispondenza ridurrà notevolmente l'efficienza di estensione dei frammenti. Pertanto, la normale Taq polimerasi non può amplificare efficacemente i frammenti bersaglio più grandi di 5 kb. La Taq polimerasi con modifica speciale o altra polimerasi ad alta fedeltà dovrebbe essere selezionata per migliorare l'efficienza dell'estensione e soddisfare le esigenze di amplificazione di frammenti lunghi. Inoltre, l'amplificazione di frammenti lunghi richiede anche la corrispondente regolazione del disegno del primer, del tempo di denaturazione, del tempo di estensione, del pH del tampone, ecc. Di solito, i primer con 18-24 bp possono portare a una resa migliore. Per evitare danni al templato, il tempo di denaturazione a 94 °C deve essere ridotto a 30 secondi o meno per ciclo e il tempo per aumentare la temperatura a 94 °C prima dell'amplificazione deve essere inferiore a 1 minuto. Inoltre, l'impostazione della temperatura di estensione a circa 68°C e la progettazione del tempo di estensione in base alla velocità di 1 kb/min possono garantire un'efficace amplificazione di frammenti lunghi.
Il tasso di errore dell'amplificazione PCR può essere ridotto utilizzando varie DNA polimerasi ad alta fedeltà. Tra tutte le Taq DNA polimerasi trovate finora, l'enzima Pfu ha il più basso tasso di errore e la più alta fedeltà (vedi tabella allegata). Oltre alla selezione degli enzimi, i ricercatori possono ridurre ulteriormente il tasso di mutazione della PCR ottimizzando le condizioni di reazione, compresa l'ottimizzazione della composizione del tampone, la concentrazione della polimerasi termostabile e l'ottimizzazione del numero del ciclo della PCR.
Categorie di prodotti
PERCHÉ SCEGLIERE NOI
Dalla sua istituzione, la nostra fabbrica ha sviluppato prodotti di prima classe mondiale con l'adesione al principio
di qualità prima di tutto. I nostri prodotti hanno guadagnato un'eccellente reputazione nel settore e preziosa fiducia tra i clienti vecchi e nuovi.








